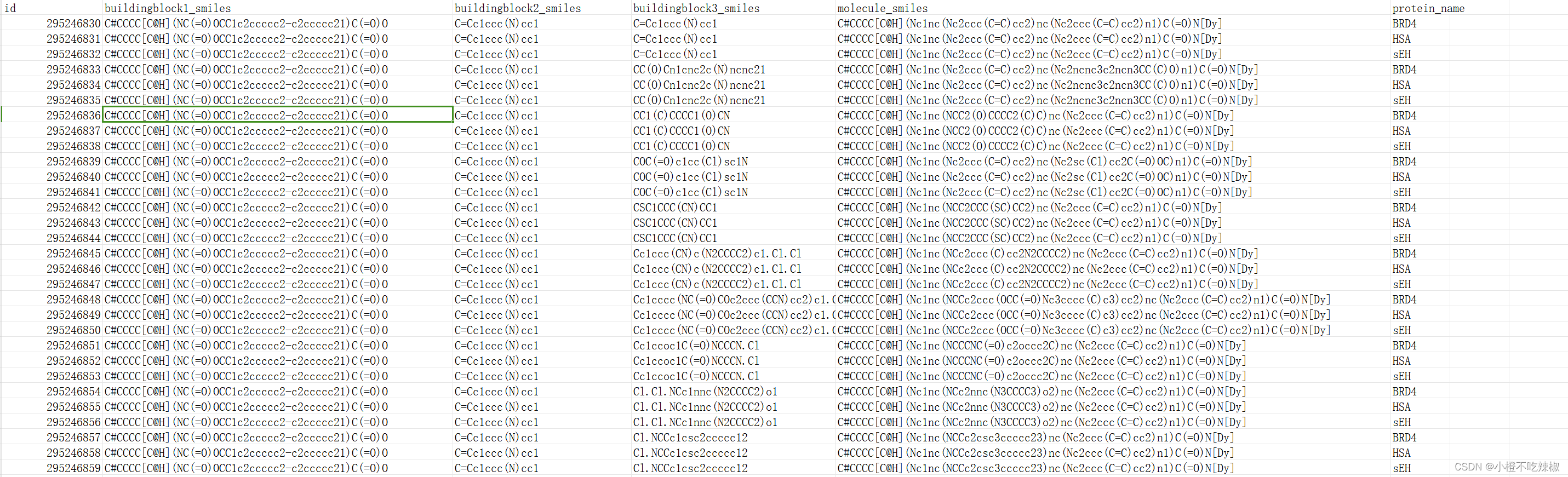
我们以Kaggle比赛里面的一个数据集跟一个公开代码为例去解释我们的OneHot编码。
简单来说,独热编码是一种将类别型变量转换为二进制表示的方法,其中每个类别被表示为一个向量,向量的长度等于类别的数量,其中只有一个元素为1,其余元素为0。例如,如果有三个类别(A、B、C),则独热编码可能如下所示:
- A: [1, 0, 0]
- B: [0, 1, 0]
- C: [0, 0, 1]
独热编码的主要优点是它将类别之间的关系消除,使得数据更适合用于机器学习算法,因为它避免了算法误认为类别之间存在顺序或距离关系。
训练集

测试集
1、导入我们相应的包
!pip install rdkitRDKit是一个开源的化学信息学工具包,用于分子建模和化学信息处理。它提供了一系列功能强大的工具,可以用于分子描述符计算、药物设计、虚拟筛选、化学信息的可视化等任务。
!pip install duckdbDuckDB是一个嵌入式的SQL数据库管理系统(DBMS),旨在提供高性能的数据查询和分析。它主要用于处理大规模数据集和分析任务,并且可以与现有的数据科学工具和应用程序集成。
2、数据准备
import duckdb
import pandas as pd
train_path = '/kaggle/input/leash-predict-chemical-bindings/train.parquet'
test_path = '/kaggle/input/leash-predict-chemical-bindings/test.parquet'
con = duckdb.connect()
df = con.query(f"""(SELECT *
FROM parquet_scan('{train_path}')
WHERE binds = 0
ORDER BY random()
LIMIT 30000)
UNION ALL
(SELECT *
FROM parquet_scan('{train_path}')
WHERE binds = 1
ORDER BY random()
LIMIT 30000)""").df()
con.close()
- 这部分代码连接到了一个DuckDB数据库,并从训练数据的parquet文件中获取数据。它选择了相等数量的绑定(binds=1)和非绑定(binds=0)的样本,以避免模型对某一类别的偏好。
- 查询语句将绑定为0和绑定为1的样本合并到一个DataFrame中,每个类别各30000个样本。最终的DataFrame包含了分子数据以及其对应的标签。
con.query()用于执行 SQL 查询,该查询从指定的 .parquet 文件中检索数据。con.close()是用于关闭与数据库的连接,它的作用是释放资源并断开与数据库的通信连接。在使用数据库时,连接是有限资源,因此在不再需要连接时应该显式地关闭它,以释放资源并避免资源泄露。
2、特征预处理
-
from rdkit import Chem from rdkit.Chem import AllChem from sklearn.model_selection import train_test_split from sklearn.metrics import average_precision_score from sklearn.preprocessing import OneHotEncoder import xgboost as xgb # Convert SMILES to RDKit molecules df['molecule'] = df['molecule_smiles'].apply(Chem.MolFromSmiles) # Generate ECFPs def generate_ecfp(molecule, radius=2, bits=1024): if molecule is None: return None return list(AllChem.GetMorganFingerprintAsBitVect(molecule, radius, nBits=bits)) df['ecfp'] = df['molecule'].apply(generate_ecfp) -
这部分代码使用RDKit库将SMILES字符串转换为RDKit的分子对象,并定义了一个函数generate_ecfp来生成ECFP特征。
-
generate_ecfp函数计算了每个分子的ECFP特征,并将其作为新的特征列添加到DataFrame中。
3、模型训练
from sklearn.ensemble import RandomForestClassifier
from sklearn.model_selection import train_test_split
# One-hot encode the protein_name
onehot_encoder = OneHotEncoder(sparse_output=False)
protein_onehot = onehot_encoder.fit_transform(df['protein_name'].values.reshape(-1, 1))
# Combine ECFPs and one-hot encoded protein_name
X = [ecfp + protein for ecfp, protein in zip(df['ecfp'].tolist(), protein_onehot.tolist())]
y = df['binds'].tolist()
# Split the data into train and test sets
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=42)
# Create and train the random forest model
rf_model = RandomForestClassifier(n_estimators=100, random_state=42)
rf_model.fit(X_train, y_train)
- 这部分代码使用随机森林分类器对数据进行训练。首先,使用
fit_transform方法对DataFrame中的'protein_name'列进行编码,并将结果存储在protein_onehot中。对蛋白质名称进行了独热编码。.values.reshape(-1, 1):这部分代码将选定的列中的数据转换为 NumPy 数组,并对其进行重塑,将其变成一个列向量。.values将 DataFrame 列转换为 NumPy 数组,.reshape(-1, 1)将数组重塑为一个列向量,其中-1表示未知的行数,而1表示只有一列。 - 通过使用列表推导式,将每个ECFPs特征向量和对应的独热编码蛋白质名称逐一组合,并将结果存储在
X中,作为模型的输入特征。 - 训练数据被划分为训练集和验证集,并使用随机森林模型进行训练。
random_state=42用于设置随机种子,保证划分的结果可以重现。
4、模型评估
from sklearn.metrics import average_precision_score
# Make predictions on the test set
y_pred_proba = rf_model.predict_proba(X_test)[:, 1] # Probability of the positive class
# Calculate the mean average precision
map_score = average_precision_score(y_test, y_pred_proba)
print(f"Mean Average Precision (mAP): {map_score:.2f}")
- 这部分代码使用训练好的随机森林模型在测试集上进行预测,得到了每个样本属于正类的概率,并将结果存储在
y_pred_proba中。 - 这部分代码用测试集上的预测结果评估了模型的性能。使用平均精度(Average Precision)评估模型在验证集上的性能。
5、测试预测
import os
# Process the test.parquet file chunk by chunk
test_file = '/kaggle/input/leash-predict-chemical-bindings/test.csv'
output_file = 'submission.csv'
# Read the test.parquet file into a pandas DataFrame
for df_test in pd.read_csv(test_file, chunksize=100000):
# Generate ECFPs for the molecule_smiles
df_test['molecule'] = df_test['molecule_smiles'].apply(Chem.MolFromSmiles)
df_test['ecfp'] = df_test['molecule'].apply(generate_ecfp)
# One-hot encode the protein_name
protein_onehot = onehot_encoder.transform(df_test['protein_name'].values.reshape(-1, 1))
# Combine ECFPs and one-hot encoded protein_name
X_test = [ecfp + protein for ecfp, protein in zip(df_test['ecfp'].tolist(), protein_onehot.tolist())]
# Predict the probabilities
probabilities = rf_model.predict_proba(X_test)[:, 1]
# Create a DataFrame with 'id' and 'probability' columns
output_df = pd.DataFrame({'id': df_test['id'], 'binds': probabilities})
# Save the output DataFrame to a CSV file
output_df.to_csv(output_file, index=False, mode='a', header=not os.path.exists(output_file))
这部分代码用训练好的随机森林模型对测试数据进行预测,并将结果保存到CSV文件中。
代码地址:Leash Tutorial - ECFPs and Random Forest | Kaggle