目录
ROC基础
生成模拟数据
率的计算
R语言计算测试
ROCR:
pROC
ROC绘制
单个ROC
两个ROC
Logistic回归的ROC曲线
timeROC
ROC基础
ROC曲线的横坐标是假阳性率,纵坐标是真阳性率,需要的结果是这个率表示疾病阳性的率(而不能误算为阴性的率)。
ROC阳性结果还是阴性结果?_roc曲线计算阳性阴性预测值-CSDN博客
生成模拟数据
rm(list = ls())
set.seed(1234)
#模拟数据
ca125 <- c(rnorm(10,80,20),rnorm(20,50,10))
##前10个均值为80,标准差为20的正态分布数据,后20个均值为50,标准差为10的正态分布随机数
group <- c(rep(c("肿瘤","非肿瘤"),c(10,20)))#
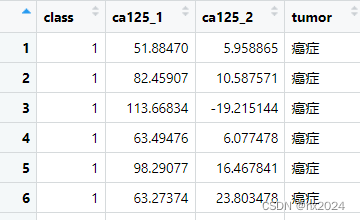
data <- data.frame(ca125,group)head(data)
ca125 group 1 55.85869 肿瘤 2 85.54858 肿瘤 3 101.68882 肿瘤 4 33.08605 肿瘤 5 88.58249 肿瘤 6 90.12112 肿瘤
率的计算
#设置ca125>60,判断为肿瘤
data$pre <- ifelse(data$ca125 > 60,"猜他是肿瘤","猜他不是肿瘤")
#列联表
table(data$group,data$pre)
猜他不是肿瘤 猜他是肿瘤
非肿瘤 19 1
肿瘤 2 8根据这个四格表我们就能算出目前的真阳性率和假阳性率:
真阳性率:猜他是肿瘤猜对的人数 / 所有肿瘤人数
假阳性率:猜他是肿瘤猜错的人数 / 所有非肿瘤人数
真阳性率 = 8 / (2+8) = 0.8
假阳性率 = 1 / (19+1) = 0.05
如果目标结果是确定非肿瘤患者:
假阳性率:猜他不是肿瘤猜错的人数 / 所有肿瘤人数
真阳性率:猜他不是肿瘤猜对的人数 / 所有非肿瘤人数
假阳性率 = 2 / (2+8) = 0.2
真阳性率 = 19 / (19+1) = 0.95
R语言计算测试
R语言计算AUC(ROC曲线)的注意事项_r语言auc-CSDN博客
ROCR:
ROCR默认计算顺序靠后的类别的AUC。如果提供给labels的值是有序因子型变量,则排在前面的默认是阴性结果(negtive),排在后面的默认是阳性结果(positive),默认计算阳性结果(排序靠后)的AUC。如果是无序因子、数值、字符、逻辑型变量,会按照R语言的默认排序,比如按照数字大小、首字母顺序等,也是计算排序靠后的类别的AUC。
set.seed(20220840)
ca125_1 <- c(rnorm(10,80,20),rnorm(20,50,10))
ca125_2 <- c(rnorm(10,20,20),rnorm(20,70,10))
class=c(rep(1:0,c(10,20)))
tumor <- c(rep(c("癌症","非癌症"),c(10,20)))
df <- data.frame(`class`=class,`ca125_1`=ca125_1,`ca125_2`=ca125_2,
`tumor`=tumor
)
library(ROCR)
pred <- prediction(predictions = ca125_1, # 预测指标
labels = tumor # 真实结果
)
performance(pred, "auc")@y.values[[1]]
# [1] 0.075这里我们想计算癌症的AUC,而不是非癌症的AUC,手动指定顺序!
pred <- prediction(predictions = ca125_1, # 预测指标
labels = tumor # 真实结果
,label.ordering = c("非癌症","癌症") # 此时就是计算癌症的AUC
)
performance(pred, "auc")@y.values[[1]]
## [1] 0.925pROC
pROC包计算AUC也需要用来预测结果的指标以及真实结果。
这个包计算pROC略有不同,它是根据中位数来的,谁的中位数大,就计算谁的AUC,比如我们的这个例子,计算下中位数看看:
ca125_1
# 把ca125_1按照tumor的两个类别进行分组,然后分别计算中位数
tapply(ca125_1, tumor, median)
## 癌症 非癌症
## 81.34426 49.99926(肿瘤的均值大)
library(pROC)
roc(response=tumor, predictor=ca125_1)
#Area under the curve: 0.925(计算结果为癌症的)ca125_2:是计算非癌症的AUC。
tapply(ca125_2, tumor, median)
## 癌症 非癌症
## 13.52771 69.69272(非肿瘤的平均数大)
roc(response=tumor, predictor=ca125_2)
Data: ca125_2 in 10 controls (tumor 癌症) < 20 cases (tumor 非癌症).
Area under the curve: 0.9
#需要设置levels和direction
# 此时计算的就是癌症的AUC
roc(response=tumor, predictor=ca125_2,
levels=c("非癌症", "癌症"), # 这个顺序随便设定,重要的是direction
direction = "<" # 手动设定非癌症 < 癌症
)
#Data: ca125_2 in 20 controls (tumor 非癌症) < 10 cases (tumor 癌症).
#Area under the curve: 0.1ROC绘制
各章示例代码/Chapter13 临床诊断实验评价.R · 杨敏迪/Analysis of Medical data by R language - 码云 - 开源中国 (gitee.com)
单个ROC
数据为动脉瘤性蛛网膜下腔出血患者的检测数据和预后:s100b是一个血清指标,outcome根据格拉斯哥评分分为good(4-5分:这里最大为5) 、poor(1-3分)

rm(list = ls())
library(pROC)
data(aSAH)
roc1 <- roc(outcome ~ s100b, data = aSAH)
attributes(roc1)#查看结果包含内容
roc1$auc#
#Area under the curve: 0.7314> #求约登指数
> roc.result <- data.frame(threshold = roc1$thresholds,
+ sensitivity = roc1$sensitivities,
+ specificity = roc1$specificities)
> View(roc.result)
> roc.result$youden <- roc.result$sensitivity + roc.result$specificity - 1
> head(roc.result)
threshold sensitivity specificity youden
1 -Inf 1.0000000 0.00000000 0.00000000
2 0.035 0.9756098 0.00000000 -0.02439024
3 0.045 0.9756098 0.06944444 0.04505420
4 0.055 0.9756098 0.11111111 0.08672087
5 0.065 0.9756098 0.13888889 0.11449864
6 0.075 0.9024390 0.22222222 0.12466125
> #找出约登指数最大的一行
> which.max(roc.result$youden)
[1] 18
> roc.result[18, ]
threshold sensitivity specificity youden
18 0.205 0.6341463 0.8055556 0.4397019绘图
?plot.roc#查看参数细节
plot.roc(roc1,
print.auc = TRUE,
auc.polygon = TRUE,
grid = c(0.1,0.2),
grid.col = c("green","red"),
auc.polygon.col = "lightblue",
print.thres = TRUE)
#AUC的置信区间——DeLong法
ci.auc(roc1)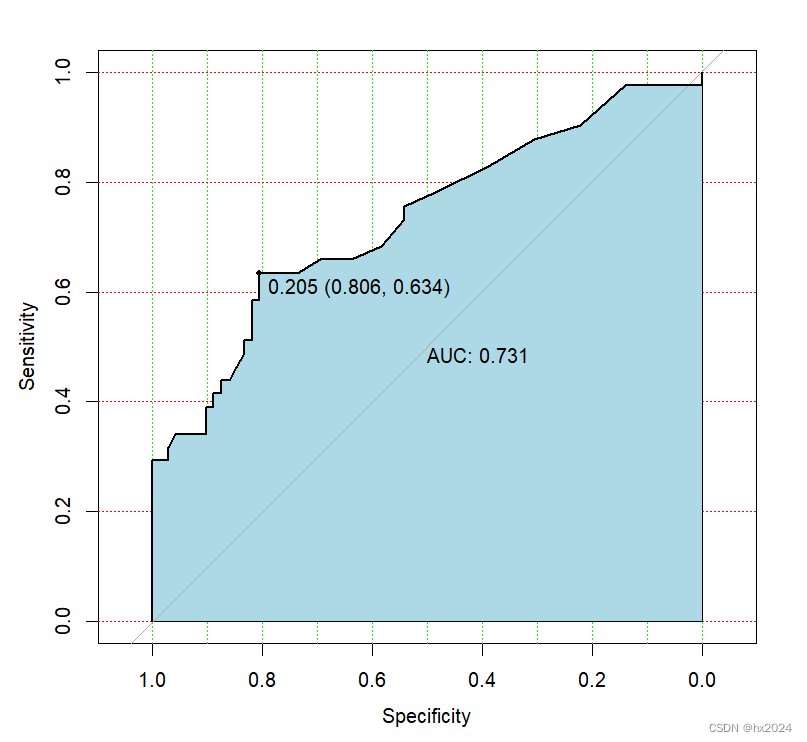
两个ROC
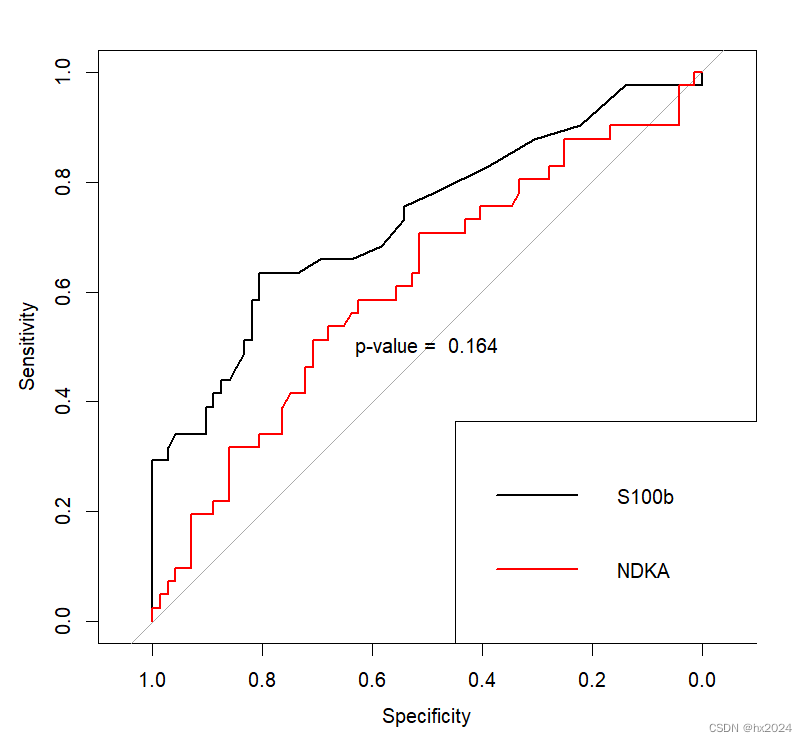
roc1 <- roc(aSAH$outcome, aSAH$s100b)
roc2 <- roc(aSAH$outcome, aSAH$ndka)
#DeLong非参数方法,Venkatraman回归模型法,bootstrap重抽样法
#默认为DeLong法
#默认为两组相关检测结果AUC的比较
#独立:参数paired = FALSE
roc.test(roc1,roc2)
#绘图
plot(roc1)
lines(roc2, col = "red")
test <- roc.test(roc1, roc2)
text(0.5,0.5, labels = paste("p-value = ",round(test$p.value, 3)))
legend("bottomright",
legend = c("S100b", "NDKA"),
col = c("1","red"), lwd = 2)
Logistic回归的ROC曲线
二分类变量

data = infert
#建立Logistic回归模型
fit <- glm(case ~ induced + spontaneous, family = binomial, data = infert)
library(epiDisplay)
logistic.display(fit)
#ROC绘制
lroc(fit, line.col = "red", lwd = 2)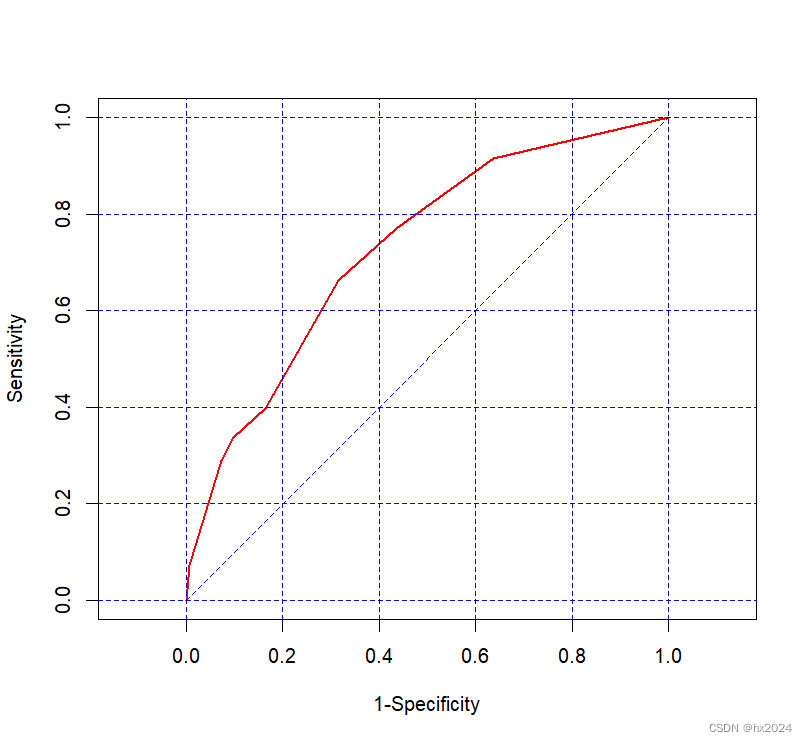
timeROC
如何绘制时间依赖性ROC曲线? (qq.com)
参考:
1:《R语言医学数据分析实战》
2:R语言计算AUC(ROC曲线)的注意事项_r语言auc-CSDN博客
3:ROC阳性结果还是阴性结果?_roc曲线计算阳性阴性预测值-CSDN博客
4:各章示例代码/Chapter13 临床诊断实验评价.R · 杨敏迪/Analysis of Medical data by R language - 码云 - 开源中国 (gitee.com)
5:如何绘制时间依赖性ROC曲线? (qq.com)