下面所述比较官方的内容都来自官方文档
Task2:赛题深入解析 - 飞书云文档 (feishu.cn)
赛题背景
-
强调了人工智能在科研领域,特别是在化学和药物研发中的重要性。
-
指出了PROTACs技术在靶向降解目标蛋白质方面的潜力。
赛题任务
-
要求选手使用提供的demo数据集,这个数据集包含了多个与PROTACs降解能力相关的字段。
-
允许选手通过数据增强或自行搜集数据来扩充数据集,并自行划分数据集用于训练和验证模型。
赛题提供的数据比较有限,仅有几百条,如果能搜索出合适的数据扩充数据集将会是很大的提升,当然数据的搜索也是非常困难的。
数据字段理解
赛题介绍部分并没有给出关于数据字段的具体介绍,所以这里对每个数据字段进行解释:
-
数据集包含了多个字段,如
uuid(唯一标识符)、Label(降解能力的标签,0表示降解能力较差,1表示降解能力好)、Uniprot、Target、E3 ligase、PDB等,这些都是与PROTACs分子相关的信息。 -
还包括了降解能力相关的数值指标,如
DC50(半数降解浓度)、Dmax(最大降解效率)、IC50(半抑制浓度)、EC50(半有效浓度)等。 -
还包括了与分子物理化学性质相关的字段,如
Molecular Weight(分子量)、Exact Mass(精确质量)、XLogP3(预测的脂水分配系数)等。
具体说明如下:
-
UUID: 一个唯一的标识符,用于识别数据记录。
-
Label: 可能指的是化合物或数据集的标签或名称。
-
Uniprot: 一个蛋白质数据库,提供关于蛋白质序列、结构和功能的信息。
-
Target: 目标蛋白,即PROTACs设计来降解的特定蛋白质。
-
E3 ligase: E3连接酶,一种在泛素化过程中扮演角色的酶,帮助标记蛋白质以供降解。
-
PDB: 蛋白质数据银行,一个包含蛋白质和核酸结构的数据库。
-
Name: 化合物的名称。
-
Smiles: 一种表示分子结构的字符串格式。
-
DC50 (nM): 半最大降解浓度,即达到最大降解效果一半时的化合物浓度。
-
Dmax (%): 最大降解效率,表示化合物能实现的最大降解效果的百分比。
-
Assay: 实验方法,这里可能指的是用于测定DC50、Dmax等的实验。
-
Percent degradation: 分子降解的百分比。
-
IC50 (nM, Protac to Target): 半最大抑制浓度,针对PROTACs与其目标蛋白的复合物。
-
EC50 (nM, Protac to Target): 半最大有效浓度,即达到一半最大生物效应的浓度。
-
Kd (nM, Protac to Target): 解离常数,表示PROTACs与其目标蛋白复合物的结合强度。
-
Ki (nM, Protac to Target): 抑制常数,用于描述抑制剂与酶的结合强度。
-
delta G, delta H, -T*delta S: 分别表示结合反应的吉布斯自由能变化、焓变和熵变,这些热力学参数用于描述分子间相互作用的能量状态。
-
kon, koff: 分别表示分子结合和解离的速率常数。
-
t1/2 (s, Protac to Target): 半衰期,即分子浓度减少到初始浓度一半所需的时间。
-
Assay (Protac to Target, kon/koff/t1/2): 可能是指测定结合和解离速率常数以及半衰期的实验方法。
接下来的部分讨论了与E3连接酶的相互作用,以及形成的三元复合物(目标蛋白、PROTACs、E3连接酶)的类似参数。
-
IC50, EC50, Kd, Ki: 与三元复合物相关的参数,与前面提到的类似,但这里特指三元复合物。
-
delta G, delta H, -T*delta S: 描述三元复合物的热力学参数。
-
kon, koff, t1/2: 描述三元复合物的动力学参数。
最后,描述了与细胞活性相关的参数,包括:
-
IC50, EC50, GI50, ED50, GR50: 这些参数描述了化合物在细胞水平上的活性,如半最大抑制浓度、半最大效应浓度等。
-
PAMPA Papp: 一种评估药物分子在仿生膜上的通透性的方法。
-
Caco-2 A2B/B2A Papp: 使用Caco-2细胞系评估药物分子的肠道吸收能力。
-
Article DOI: 文章的数字对象标识符,用于唯一标识科学文献。
-
Molecular Weight, Exact Mass: 分子量和精确质量。
-
XLogP3: 预测的辛醇/水分配系数,用于预测脂溶性。
-
Heavy Atom Count, Ring Count: 重原子数和环的数量。
-
Hydrogen Bond Acceptor/Donor Count: 氢键受体/供体计数。
-
Rotatable Bond Count: 可旋转键的数量。
-
Topological Polar Surface Area: 拓扑极性表面积。
-
Molecular Formula, InChI, InChI Key: 分子式、国际化学标识符及其密钥。
其中将对Smiles、Assay (DC50/Dmax)、Assay (Protac to Target, IC50)、Assay (Cellular activities, IC5、Article DOI、InChI展开介绍。
Smiles结构如下:
NC1=NC=NC2=C1C(C1=CC=C(OC3=CC=CC=C3)C=C1)=NN2[C@@H]1CCCN(C(=O)C2=CN(CCOCCOCCOCCCC3=CC=CC4=C3CN(C3CCC(=O)NC3=O)C4=O)N=N2)C1Smiles是一种用于描述化学结构的文本字符串,它能够被用于输入化学信息学软件。
这个特定的Smiles字符串代表了一个含有多个环和官能团的有机分子。下面是这个分子结构的一些特征:
-
NC1=NC=NC2:表示一个含氮的六元环结构,可能是一个吡啶环。 -
C1C(C1=CC=C(OC3=CC=CC=C3)C=C1):表示一个苯环(OC3=CC=CC=C3),该苯环通过一个碳原子连接到另一个苯环。 -
=NN2:表示一个氮氮双键。 -
[C@@H]1CCCN:表示一个手性中心,碳原子1连接到一个氮原子,并且有一个手性标记@@H。 -
(C(=O)C2=CN(CCOCCOCCOCCCC3=CC=CC4=C3CN(C3CCC(=O)NC3=O)C4=O)N=N2):这是一个复杂的部分,包含一个羰基(C(=O)),一个烯醇胺结构,以及多个碳链和连接的氮原子。
这个Smiles字符串代表的分子可能是一种生物活性分子,例如一种药物或生物分子。由于其复杂性,通常需要专业的化学信息学软件来解析和可视化这种结构。
Assay (DC50/Dmax)结构如下:
'Degradation of IRAK4 in HEK293T cells after 24 h treatment' 'Degradation of HDAC3 in MDA-MB-468 cells after 14 h treatment'在生物测定和药物发现领域,"Degradation of IRAK4 in HEK293T cells after 24 h treatment" 和 "Degradation of HDAC3 in MDA-MB-468 cells after 14 h treatment" 描述了两种不同的实验情境。下面是对这些信息的解释:
-
Degradation of IRAK4 in HEK293T cells after 24 h treatment:
-
这指的是在实验中,IRAK4(白细胞介素-1受体相关激酶4)在HEK293T细胞系中的降解情况。HEK293T是一种常用的人类胚胎肾细胞系,经常用于分子生物学和细胞生物学实验。
-
"24 h treatment" 表示这些细胞在实验中被处理(可能是某种药物或化合物)持续了24小时。
-
-
Degradation of HDAC3 in MDA-MB-468 cells after 14 h treatment:
-
这描述了HDAC3(组蛋白去乙酰化酶3)在MDA-MB-468细胞系中的降解情况。MDA-MB-468是一种人类乳腺癌细胞系。
-
"14 h treatment" 表示这些细胞在实验中被处理持续了14小时。
-
-
Assay (DC50/Dmax):
-
这可能指的是一种实验测定方法,用于评估化合物对蛋白质降解的影响。
-
"DC50" 可能是指半最大降解浓度(Degradation Concentration 50),即导致蛋白质降解达到最大值一半的化合物浓度。
-
"Dmax" 可能是指在实验条件下可观察到的最大降解效果。
-
"Assay (DC50/Dmax)" 可能是一个比率或关系,用来量化化合物诱导的蛋白质降解效率。
-
在药物筛选和细胞生物学实验中,这类信息对于评估化合物的潜在药效和毒性至关重要。通过测量特定蛋白质的降解情况,研究人员可以了解化合物对细胞功能的影响,并进一步研究其作用机制。
InChI结构如下:
InChI=1S/C47H61N7O6S/c1-7-26-49-53-44(58)36-20-16-33(17-21-36)34-22-24-37(25-23-34)51-40(56)12-10-8-9-11-13-41(57)52-43(47(4,5)6)46(60)54-28-38(55)27-39(54)45(59)50-30(2)32-14-18-35(19-15-32)42-31(3)48-29-61-42/h14-25,29-30,38-39,43,49,55H,7-13,26-28H2,1-6H3,(H,50,59)(H,51,56)(H,52,57)(H,53,58)/t30-,38+,39-,43+/m0/s1InChI(国际化学标识符)是一种用于唯一标识化学化合物的标准化字符串。它由一系列部分组成,提供了关于分子结构的详细信息。下面是对提供的InChI字符串的详细解释:
-
开头标识:
-
InChI=1S/开头的1S表示这是一个标准InChI字符串。
-
-
分子式:
-
C47H61N7O6S表示该化合物的分子式,包含47个碳原子(C)、61个氢原子(H)、7个氮原子(N)、6个氧原子(O)和1个硫原子(S)。
-
-
连接表:
-
/c1-7-26-49-53-44(58)36-20-16-33(17-21-36)34-22-24-37(25-23-34)51-40(56)12-10-8-9-11-13-41(57)52-43(47(4,5)6)46(60)54-28-38(55)27-39(54)45(59)50-30(2)32-14-18-35(19-15-32)42-31(3)48-29-61-42/这部分是连接表,描述了原子在分子中的连接顺序和方式。
-
-
氢原子计数:
-
h14-25,29-30,38-39,43,49,55H表示在这些碳原子上附加的氢原子数量。
-
-
多可旋转键计数:
-
7-13,26-28H2表示在这些碳原子之间有两个氢原子,可能形成可旋转键。
-
-
立体化学信息:
-
(H,50,59)(H,51,56)(H,52,57)(H,53,58)表示分子中存在多个手性中心,每个手性中心由一对氢原子标记。
-
-
同分异构体信息:
-
/t30-,38+,39-,43+表示分子中某些碳原子的立体化学构型,如30-表示一个向下的楔形表示的碳原子,38+表示一个向上的楔形表示的碳原子。
-
-
混合物或互变异构体信息:
-
/m0表示这是一个单一的化合物,没有互变异构体。
-
-
电荷和自旋多重度信息:
-
/s1表示分子的电荷和自旋多重度信息,这里s1表示单线态。
-
-
结束标识:
-
InChI字符串以
/结尾。
-
InChI提供了一种非常详细的化学结构表示方法,使得不同的化学信息学软件能够准确无歧义地解析和理解分子结构。通过InChI,研究人员可以确保在不同的数据库和软件平台之间准确交换化学结构信息。
预测目标
-
选手需要预测PROTACs的降解能力,具体来说,就是预测
Label字段的值。 -
根据
DC50和Dmax的值来判断降解能力的好坏:如果DC50大于100nM且Dmax小于80%,则Label为0;如果DC50小于等于100nM或Dmax大于等于80%,则Label为1。
参考资料
-
药物化学与PROTACs相关文献:
-
"Targeted Protein Degradation by Small Molecules" (综述PROTACs的原理和应用)
-
"The Role of E3 Ligases in Targeted Protein Degradation" (关于E3连接酶在蛋白质降解中的作用)
-
-
生物信息学与化学信息学资源:
-
RDKit官方文档:一个开源化学信息学软件库,用于处理化学分子和相关数据。http://rdkit.chenzhaoqiang.com/index.html
-
Biopython官方文档:用于生物计算的Python库。https://biopython-cn.readthedocs.io/zh-cn/latest/
-
其中RDKit库可以帮助处理SMILES字符串,如下代码可以将SMILES转换为分子对象,然后进行分子结构绘制
from rdkit import Chem # 导入Chem模块,它包含了处理分子表示的功能。
from rdkit.Chem import AllChem, Draw # 从Chem模块中导入AllChem和Draw子模块。AllChem提供了额外的化学功能,如2D和3D坐标生成、分子性质计算等。Draw模块则用于绘制分子结构图。
# SMILES字符串
smiles = "NC1=NC=NC2=C1C(C1=CC=C(OC3=CC=CC=C3)C=C1)=NN2[C@@H]1CCCN(C(=O)C2=CN(CCOCCOCCOCCCC3=CC=CC4=C3CN(C3CCC(=O)NC3=O)C4=O)N=N2)C1" # SMILES(Simplified Molecular Input Line Entry System)是一种用于描述化学物质结构的简化线性文本表示法。
# 3 将SMILES转换为分子对象
mol = Chem.MolFromSmiles(smiles) # 将SMILES字符串转换成RDKit中的Mol对象,这是RDKit中表示分子的核心数据结构。
# 4检查分子是否有效
if mol is None:
print("The SMILES string is invalid.")# 如果转换失败,mol将会是None。这种情况下,代码会打印出"The SMILES string is invalid."(SMILES字符串无效)
else:
print("The molecule was parsed successfully.") #如果转换成功,代码将打印出"The molecule was parsed successfully."(分子已成功解析)。
# 绘制分子结构
molDrawer = Draw.MolToMPL(mol) # 使用Draw模块中的MolToMPL函数来将分子对象转换为matplotlib图形对象。
molDrawer.show() # 显示生成的分子结构图。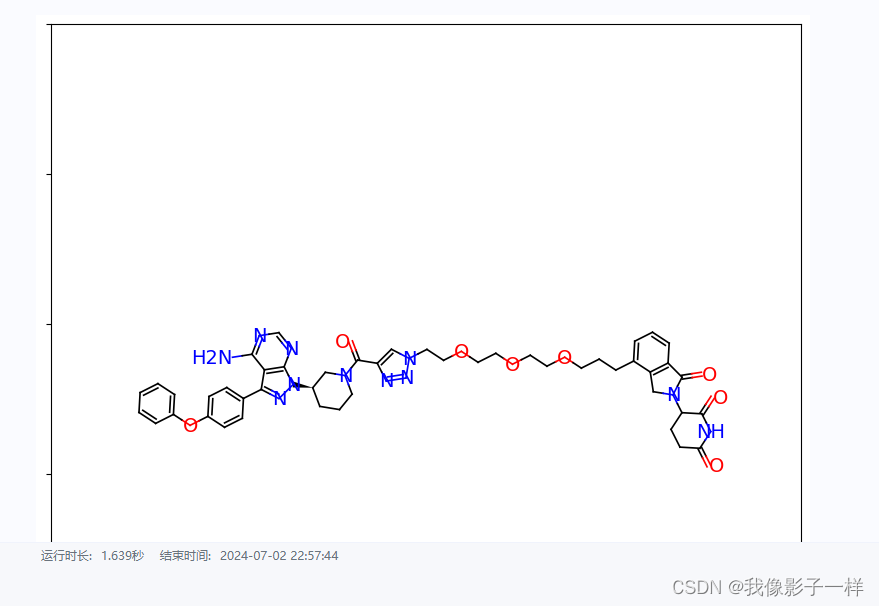
个人感悟
- 上面内容从头看到尾,字都认识,但是由于术语太专业,合起来就看不懂了
- 大致了解了所有子段的意思,明白了上次的运行结果文件里的字段label里面的0和1的意思
- 应该是基于其他字段的信息构建模型,从而预测PROTACs的降解目标蛋白质方面的潜力
- 感觉还是需要化学生物学方面的知识